
Mesurer, à partir de modèles moléculaires, l'épaisseur d'un paire de nucléotides.
Thème pédagogique : La réplication de l'ADN
Niveau : Première spécialité SVT
Connaissance :
Chaque chromatide est constituée d'une longue molécule d'ADN associée à des protéines structurantes.
Capacités :
Calculer la longueur totale d’une molécule d’ADN dans un chromosome et de l’ensemble de l’ADN d’une cellule humaine ; comparer avec le diamètre d’une cellule. Calculer la longueur d’ADN de l’ensemble des cellules humaines.
Les modèles moléculaires disponibles dans les banques de données, comme la PDB (Protein Data Bank), décrivent la position des atomes dans les molécules. Les visualisateurs, comme Libmol, permettent d'afficher la structure de la molécule et d'y effectuer des mesures. On cherche ici à déterminer l’épaisseur une paire de nucléotides dans l'ADN pour disposer de la taille d'un élément constitutif de l'ADN.
1. Mesure de l’épaisseur d’un nucléotide
Détermination à partir de modèles moléculaires de l’épaisseur d’une paire de nucléotide.
Différents modèles moléculaires permettent l'activité. Les deux proposé ci-dessous ou encore les modèles 1BNA ; 1AOI, 5OY7.
Exploiter les fonctionnalités du logiciel Libmol pour déterminer la taille (épaisseur) d’une paire de nucléotides.
|
Épaisseur d’une paire de nucléotide (nm) :
|
|
Aide à la réalisation :
UTILISER l'outil de MESURE de Libmol : 
Réaliser les MESURES utiles.
Éléments de réponse :
Détermination de l’épaisseur d’une paire de nucléotide avec Libmol.
Deux méthodes peuvent permettre de mesurer l'épaisseur d'une paire de bases. Elles reposent sur la recherche d'éléments répétitifs le long de la molécule. Cela peut être des atomes de références entre deux nucléotides. Par exemple
Méthode 1 : Utilisation d’atomes de référence pour trouver l’épaisseur d'une paire de nucléotides.
On mesure :
- soit la distance entre les atomes extrêmes d'un nucléotide : de C5' à O3' d'un nucléotide (DT7 dans l'exemple ci-dessous). On mesure ici l'épaisseur d'un nucléotide.
- soit la distance entre deux atomes superposés d'une séquence de nucléotide : O4 d'un nucléotide N à C5 du nucléotide N+2 (DT8 à DA6 dans l'exemple ci-dessous). On mesure ici l'épaisseur de demi nucléotide et d'un nucléotide complet soit 2 nucléotides (1/2 N1 + N2 + 1/2 N3 ).
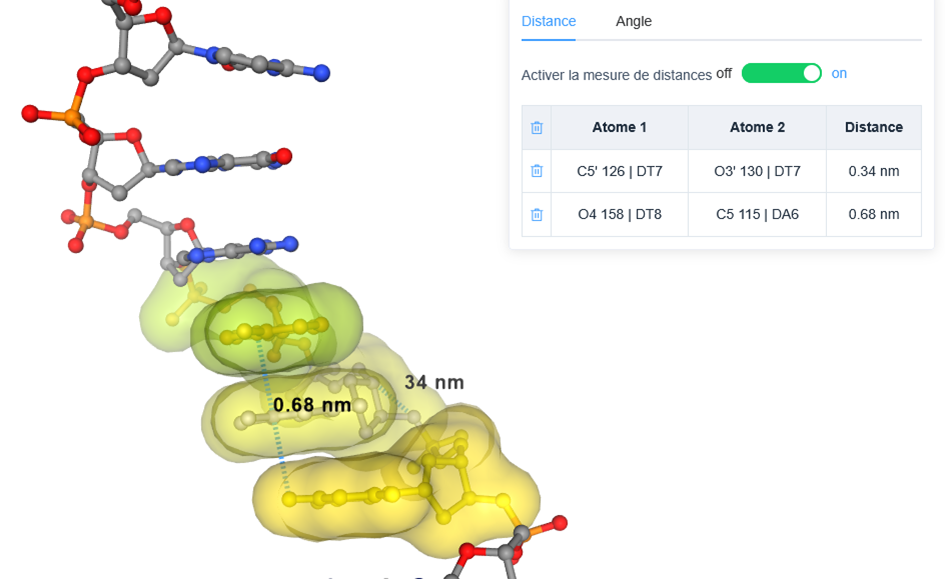
Dans ces exemples on trouve :
- soit 0,34 nm qui correspond à l'épaisseur d'un nucléotide,
- soit 0,68 nm qui correspond à l'épaisseur de deux nucléotides soit 0,68/2 = 0,34 nm pour un nucléotide.
Méthode 2 : Utilisation d’un pas de la double hélice d’ADN pour trouver l’épaisseur d’un nucléotide.
On utilise les fonctionnalités d'affichage de Libmol pour repérer un pas d'hélice dans l'ADN c'est à dire la superposition exacte de deux nucléotides après un tour complet. La séquence permet de constater que cela correspond 11 nucléotides soit l'épaisseur de 10 nucléotides (1/2N1 + 1*(N2 à N10) +1/2N11).
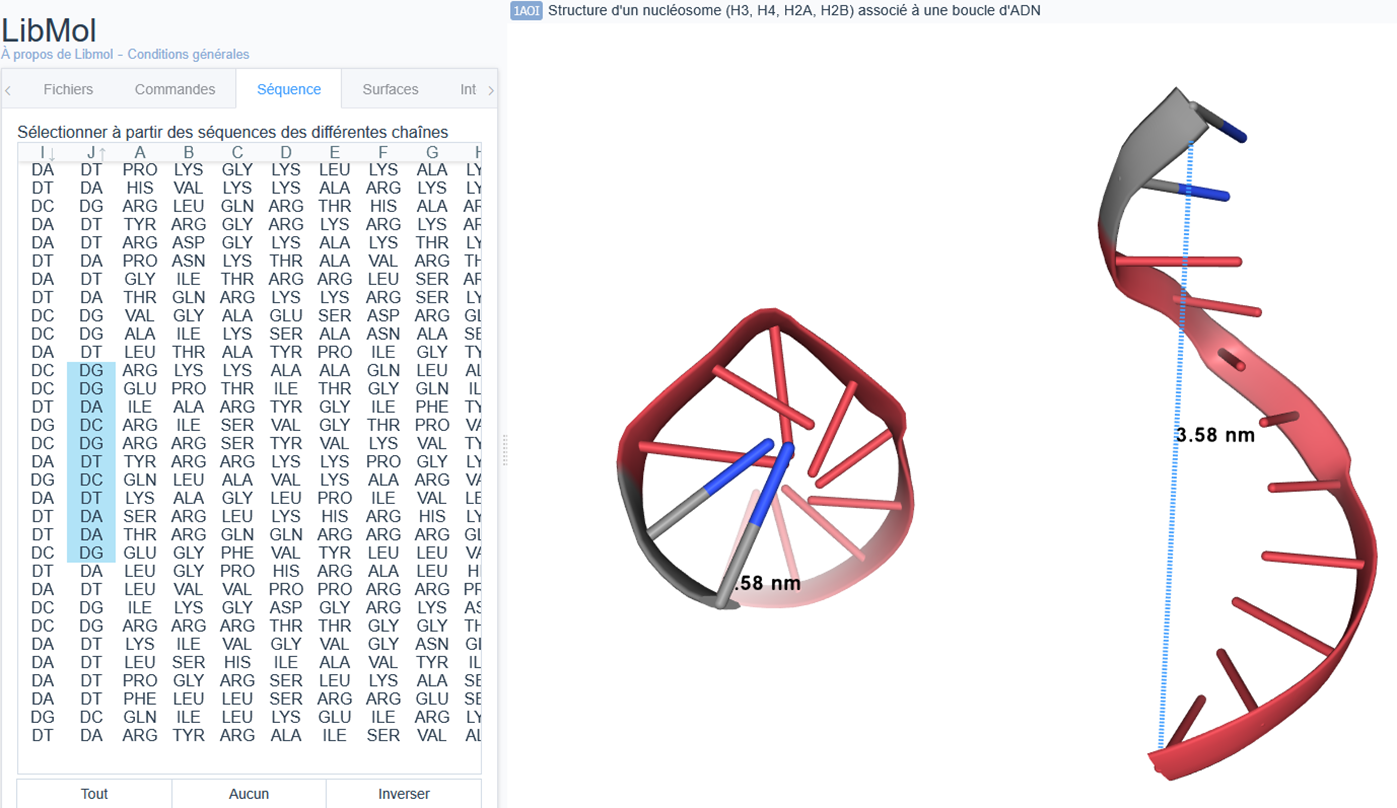
Dans cet exemple, on trouve 3,58 nm qui correspond à l'épaisseur de 10 nucléotides soit 3,58/10 = 0,36 nm pour un nucléotide.
ou encore :
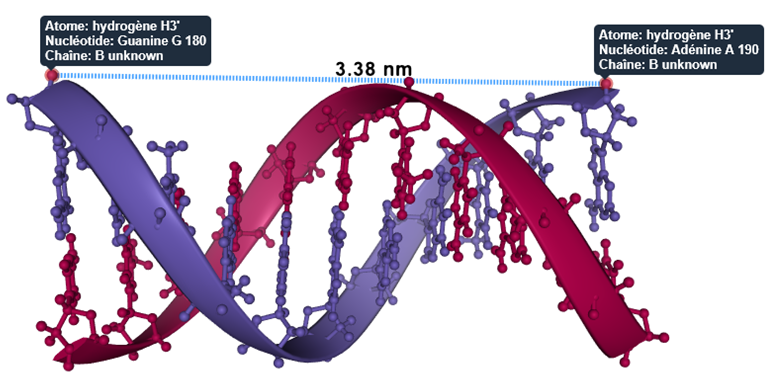
Dans cet exemple, on trouve 3,38 nm qui correspond à l'épaisseur de 10 nucléotides soit 3,38/10 = 0,34 nm pour un nucléotide.
Des exemples qui permettent de discuter la notion d'incertitude des mesures.



